


Bien que les modifications chimiques de l’ARN soient connues depuis les années 1970, les avancées en spectrométrie de masse et en séquençage de nouvelle génération (NGS) ont révélé leur importance biologique. Ces marques constituent une nouvelle variable épigénétique essentielle à la régulation de l’expression génique (épissage, stabilité, traduction…) et des fonctions biologiques majeures. Plus de 150 modifications de l’ARN ont été identifiées à ce jour, reflétant un métabolisme complexe et dynamique, indicateur en temps réel de l’état cellulaire ou tissulaire. Au-delà de leur rôle fonctionnel, elles ouvrent des perspectives prometteuses en médecine personnalisée, notamment pour le diagnostic et la prise en charge des maladies. La dérégulation des effecteurs de l’épigénétique de l’ARN — classés en "writers", "erasers" et "readers" — est liée au développement de pathologies humaines, comme le cancer, favorisant progression tumorale, dissémination, résistances thérapeutiques et récidives.
![]()
Axe 1 : EpiTransDiaG - Exploiter l’expitranscriptome pour le diagnostic et le dépistage du cancer (Responsables: A. David & S. Quesada)
Cet axe combine la spectrométrie de masse et l'apprentissage automatique pour convertir les informations chimiques contenues dans les échantillons d'ARN extraits de cohortes de patients atteints de cancer (tissus et biofluides) en signature(s) épitranscriptomique(s) pouvant être exploitées pour le diagnostic et le pronostic. À cette fin, un pipeline expérimental et analytique a été établi au sein d'une plateforme spécialisée dans le développement de biomarqueurs cliniques (PPC-SMART). EpiTransDiaG exploite la sensibilité extrême de la spectrométrie de masse et la puissance de l'intelligence artificielle pour prédire la présence de la maladie à partir de très petites quantités d’échantillons (deux gouttes de sang), en quelques heures, et avec une efficacité redoutable. En partenariat avec les équipes de l'ICM et du CHU, ce projet fait l'objet d'un programme de maturation SATT (2023-2025).
Trois axes de recherche sont développés par notre équipe, en synergie avec le consortium SMART, le GDR « ARN », le réseau européen TRANSLACORE et les équipes cliniques de l'ICM et du CHU.
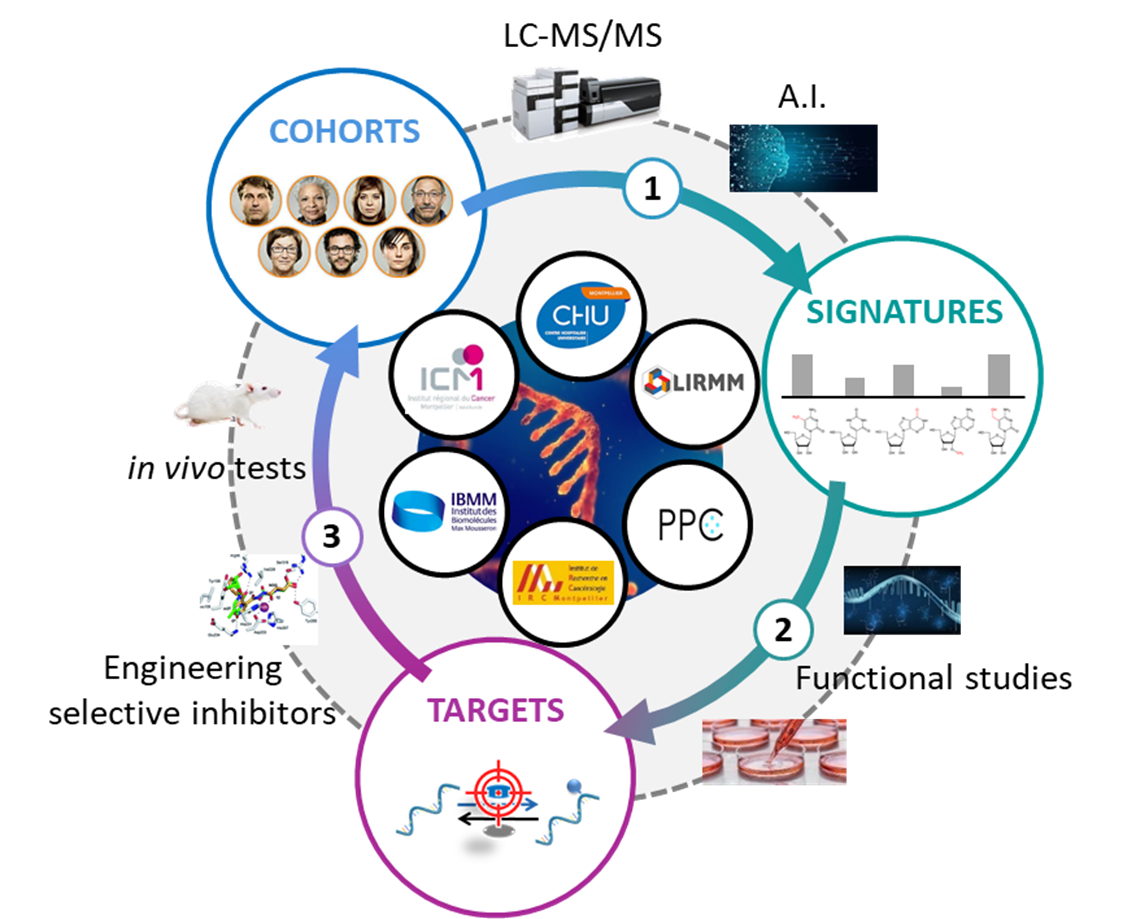
Description des axes de recherche majeurs de l’équipe (sur la version définitive, ces parties seront uniquement déroulées grâce à un bouton « + ». moins de 700 caractères par axe
Axe 2 : SCoRE – Etudier l’impact de l’épitranscriptome sur l’adaptation des cellules cancéreuses (Responsables: A. David & C. Planque)
Sur la base des données cliniques du premier axe, le deuxième axe, SCoRE, identifie des marqueurs chimiques "fonctionnellement" impliqués dans la progression/l'adaptation du cancer, dont les effecteurs enzymatiques peuvent représenter une cible thérapeutique potentielle. Deux modifications sont actuellement à l'étude. Premièrement, la N6,2′-O-diméthyladénosine (m6Am), une modification adjacente à la coiffe de l'ARNm, impliquée dans la maturation, la stabilité et le contrôle traductionnel de l'ARNm. Deuxièmement, la queuosine, un nucléoside hypermodifié d'origine bactérienne situé à la position 34 (wobble) de quatre ARNt, régulant l'efficacité (et la fidélité) de la traduction des codons correspondants.
Axe 3: EpiCure - Concevoir de nouveaux concepts thérapeutiques basés sur l'ARN et sa chimie (Responsables: A. David & F. Macari)
Le dernier axe, EpiCure, vise à modéliser et concevoir des composés thérapeutiques basés sur l’ARN, en particulier des inhibiteurs ciblant les enzymes régulant l'épitranscriptome. Cette étude est réalisée en étroite collaboration avec trois équipes de chimie (M. Duca, ICN ; M. Smietana, IBMM ; M. Etheve-Quelquejeu, LCBPT). Ce programme inclut également la conception d'ARNm thérapeutiques sur mesure dans le cadre de l'IHU IMMUN4CURE.